Organisation du noyau en quiescence
Bien que le noyau de la levure bourgeonnante ait une organisation bien définie, la plupart des études sur cette organisation ont été réalisées dans des conditions riches en nutriments. Cependant, la disponibilité des nutriments est un facteur de stress majeur qui a un impact sur l'expression des gènes, affectant constamment et fortement les fonctions cellulaires.
Dans notre laboratoire, nous permettons aux cellules de levure d'épuiser les nutriments et d'atteindre un état stationnaire. Deux populations commencent à apparaître (Allen et al., 2006), l'une constituée de grandes cellules mères avec de multiples cicatrices de bourgeons et l'autre de cellules filles denses, principalement non bourgeonnées. Dans cette deuxième population, les cellules sont caractérisées par un arrêt réversible du cycle cellulaire, une caractéristique définissant les cellules quiescentes d'une espèce à l'autre. Dans la levure bourgeonnante, ces « cellules Q » montrent une diminution de l'activité cellulaire et de l'expression des gènes, mais plus important encore, ces cellules présentent une résistance accrue au stress et peuvent survivre sans nutriments pendant plusieurs semaines, ce qui en fait un modèle approprié pour les études sur la longévité.
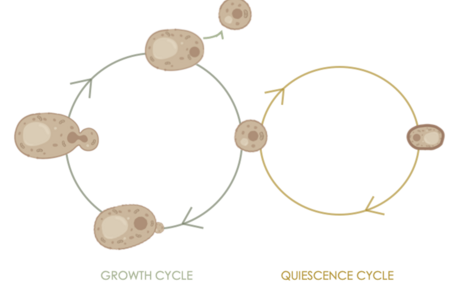
De manière frappante, les noyaux des cellules Q diffèrent par leur structure et leur fonction des noyaux des cellules cycliques. Par rapport à la phase du cycle cellulaire G1, la taille nucléaire est réduite dans les cellules Q, et tandis que les cellules de levure en cycle organisent leur génome dans une configuration de type Rabl, notre équipe a montré que les cellules Q ont leurs télomères assemblés dans un hypercluster au centre du noyau remodelant l'architecture du génome en une conformation qui pourrait contribuer à prolonger la durée de vie chronologique (CLS) des cellules quiescentes (Guidi et al., 2015). Visiblement, ces caractéristiques coïncident avec une organisation contrainte du génome, en combinaison avec une diminution massive de l'acétylation du promoteur et de l'activité transcriptionnelle (McKnight et al., 2015 ; Young et al., 2017).
Les transitions d'entrée et de sortie de cet état nous intéressent, car elles coïncident avec des réorganisations nucléaires à grande échelle et se produisent dans des échelles de temps très différentes : alors que l'ajustement de la prolifération à l'arrêt de la croissance se produit progressivement à un rythme lent (sur 5-7 jours), la sortie de la quiescence s'effectue rapidement (en quelques minutes).
Quelques-unes des pistes que nous explorons actuellement :
• Quels sont les mécanismes physiques et moléculaires qui sous-tendent la dynamique de l'architecture nucléaire en réponse aux changements des conditions environnementales ?
• Comment le reste du génome est-il organisé par rapport à l'hypercluster de télomères dans les cellules Q ?
• Où dans le génome, et dans l'espace nucléaire, se trouve la machinerie transcriptionnelle ?
• Quelle est la variabilité de cellule à cellule parmi les Q-Cells ?
• L'hétérogénéité de cellule à cellule contribue-t-elle à la capacité de la population à s'adapter à de nouvelles conditions ?
Pour répondre à ces questions, nous intégrons des données provenant de la génomique de la population entière, de la génétique, de la microscopie à fluorescence conventionnelle et des approches d'imagerie à molécule unique et à cellule unique.
Comme l'analyse de la durée de vie chronologique de la levure a souvent été prédictive des mécanismes influençant ce processus essentiel chez différentes espèces, nous prévoyons que des principes de base émergeront de nos études avec de larges implications pertinentes dans d'autres organismes.

Allen, C., Büttner, S., Aragon, A.D., Thomas, J.A., Meirelles, O., Jaetao, J.E., Benn, D., Ruby, S.W., Veenhuis, M., Madeo, F., et al. (2006). Isolation of quiescent and nonquiescent cells from yeast stationary-phase cultures. Journal of Cell Biology 174, 89–100.
Guidi, M., Ruault, M., Marbouty, M., Loïodice, I., Cournac, A., Billaudeau, C., Hocher, A., Mozziconacci, J., Koszul, R., and Taddei, A. (2015). Spatial reorganization of telomeres in long-lived quiescent cells. Genome Biol 16, 206.
McKnight, J.N., Boerma, J.W., Breeden, L.L., and Tsukiyama, T. (2015). Global Promoter Targeting of a Conserved Lysine Deacetylase for Transcriptional Shutoff during Quiescence Entry. Molecular Cell 59, 732–743.
Young, C.P., Hillyer, C., Hokamp, K., Fitzpatrick, D.J., Konstantinov, N.K., Welty, J.S., Ness, S.A., Werner-Washburne, M., Fleming, A.B., and Osley, M.A. (2017). Distinct histone methylation and transcription profiles are established during the development of cellular quiescence in yeast. BMC Genomics 18, 107.
