Réparation de l'ADN
Une variété d'agents exogènes et endogènes endommagent constamment le génome. Parmi les lésions les plus cytotoxiques et génotoxiques, les cassures double brin (DSB) ont des conséquences dramatiques pour la cellule et sa progéniture. L'incapacité à réparer ces lésions conduit soit à la mort cellulaire, soit à l'instabilité génomique et aux phénotypes des maladies qui leur sont associées, comme illustré par des organismes modèles où des mutations dans les gènes de réparation de l'ADN conduisent à une prédisposition au cancer.
Pour faire face aux DSB, les cellules mettent en place plusieurs mécanismes qui composent ensemble la réponse aux dommages à l'ADN (DDR). La DDR doit détecter, signaler et réparer les DSB. Les cellules eucaryotes ont développé deux voies de réparation principales pour réparer les DSB : la jonction d'extrémité non homologue (NHEJ) et la recombinaison homologue (HR).
Associant génétique, microscopie avancée et analyse d'images, nous étudions différentes étapes de la DDR en relation avec l'organisation nucléaire dans la levure bourgeonnante (Batté et al., 2017 ; Miné-Hattab et al., 2017) et plus récemment dans les cellules humaines. Profitant de nos découvertes selon lesquelles le répresseur Sir3 favorise la propagation de la chromatine répressive et le regroupement des télomères de manière indépendante (Ruault et al, 2011 ; voir projet 1), nous avons montré que ces deux fonctions de Sir3 contribuent à éviter la perte d'informations génétiques lors de cassures double brin subtélomériques, en favorisant des événements de recombinaison fidèle entre les sous-télomères. Ces résultats pourraient être particulièrement pertinents dans les cellules de levure quiescentes (voir projet 3).
En réponse aux cassures double brin (DSB), les protéines de réparation s'accumulent sur les sites endommagés, formant des sous-compartiments sans membrane ou "foyers". Plusieurs modèles ont été intensivement débattus dans la littérature pour expliquer leur formation, y compris la possibilité qu’ils résultent de séparation de phase liquide. Cependant, les preuves expérimentales sont souvent à la limite de la résolution optique.
En combinant la microscopie en molécules uniques et l'analyse statistique, nous avons comparé la dynamique de plusieurs protéines de réparation s'accumulant au niveau des cassures de l'ADN pour élucider leur structure, leur dynamique et leur nature physique dans les cellules vivantes.
Nous avons montré que si la plupart des molécules Rfa1 sont liées à de l’ADN simple brin, les molécules Rad52 sont libres d'explorer l'ensemble du foyer reflétant l'existence d'une gouttelette liquide autour de l’ADN endommagé (Miné-Hattab et al., 2021, Heltberg et al, 2021).
Nous développons actuellement de nouvelles approches pour visualiser les événements de recombinaison homologue clés dans les cellules vivantes, comme l’étude de la dynamique du nucléofilament formé par Rad51 sur l’ADN simple brin lors de la recherche d'homologie de séquence.
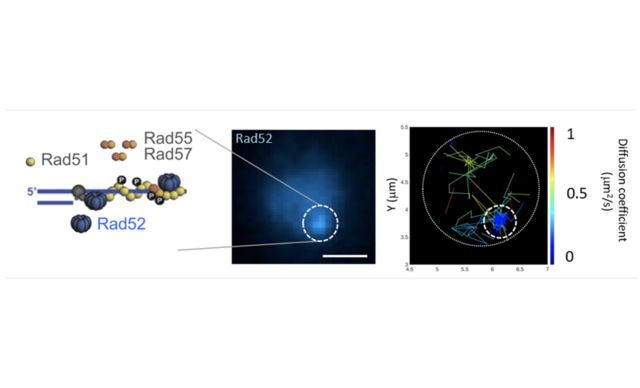
A gauche : nucléofilament réparé par recombinaison homologue. Au milieu : Foyer de réparation Rad52. A droite : Trajectoires des molécules Rad52 après cassure (SPT).
