- Accueil >
- Les actualités de l'Institut Curie >
- Transcriptomique spatiale : nouveau guide pour mieux étudier la spatialité des cellules dans les tissus
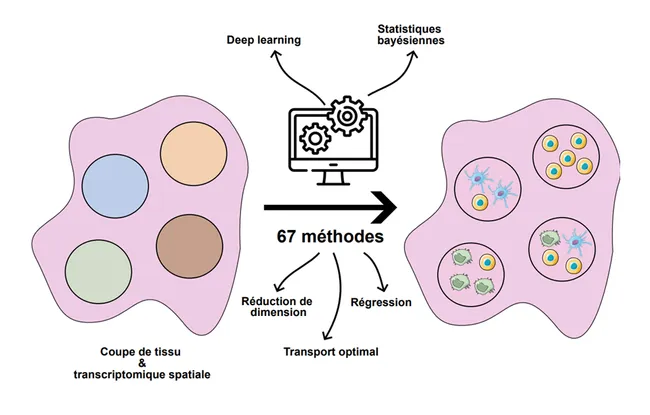
Il existe des nombreuses méthodes permettant de retrouver la distribution spatiale des cellules des tissus à partir des données de transcriptomique spatiale n’ayant pas une résolution à la cellule unique. Une équipe de l’Institut Curie, Biologie computationnelle et génomique intégrative du cancer (Inserm U1331), dirigée par le Dr Florence Cavalli, a récemment revu l’ensemble de ces méthodes d’analyse, dite de déconvolution, permettant ainsi de guider les utilisateurs vers la plus adéquate. Ce travail a fait l’objet d’une publication dans Nature Reviews Genetics le 14 mai 2025.
Les tissus multicellulaires sont constitués de différents types cellulaires, assurant leur bon fonctionnement au sein de l’organisme. Un paramètre essentiel à prendre en compte pour comprendre le fonctionnement des tissus est leur organisation spatiale, soit la localisation des différents types cellulaire au sein du tissu. En effet, ceux-ci ne sont pas forcément répartis de façon homogène et n’interagissent pas avec tous les autres types cellulaires. Dans des organes comme le cerveau, les types cellulaires comme les neurones sont précisément localisés dans différentes couches, pour assurer les fonctions de perception, ou encore la mémoire.
Il existe plusieurs méthodes pour caractériser les cellules, dont l’étude de leur expression génétique via la capture des ARNs (aussi dit transcrits), appelée la transcriptomique. De nouvelles technologies permettent désormais de caractériser ces cellules dans leur contexte spatial, c’est la transcriptomique spatiale. Cependant, la faible résolution de ces technologies rend difficile l'attribution précise des transcrits à leur cellule d'origine.
« Il existe aujourd’hui de nombreuses façons d’analyser les résultats de transcriptomique spatiale qui permettent d’associer les transcrits à leur localisation dans le tissu étudié, c’est-à-dire déterminer de quelles cellules les transcrits proviennent, par exemple. » explique Florence Cavalli.
En réalité, il existe 67 méthodes d’analyse dite de déconvolution qui permettent de décrire où les différents types cellulaires se situent dans un tissu à partir des données de transcriptomique spatiale. Ces méthodes permettent de décrire l’organisation spatiale des tissus sains mais aussi des tumeurs. Elles sont décrites et classées dans une nouvelle publication de Nature Reviews Genetics, menée par l’équipe de Florence Cavalli.
« Si quelqu’un cherche à commencer ce type d’analyse aujourd’hui, il trouvera 67 méthodes différentes. Notre travail ici a été de recenser toutes les méthodes et de résumer leurs points communs et leurs différences afin de guider l’utilisateur vers le bon choix, en fonction de la question biologique, ou de la présence potentielle d’autres données, comme celles d’imagerie, qui peuvent influencer l’analyse nécessaire. » continue Florence Cavalli.
En rassemblant et en comparant l'ensemble des méthodes d'analyse existantes, ce travail constitue un guide précieux pour orienter les utilisateurs dans le choix de l'approche la plus adaptée à leur question de recherche. Largement employées dans les domaines de la cancérologie, certaines de ces approches d’analyses de données de transcriptomique spatiales sont notamment utilisées dans le laboratoire de Dr Florence Cavalli, pour étudier le rôle des niches cellulaires dans les tumeurs cérébrales de l’adulte et de l’enfant.
Références : Lucie C. Gaspard-Boulinc, Luca Gortana, Thomas Walter, Emmanuel Barillot & Florence M. G. Cavalli. Cell-type deconvolution methods for spatial transcriptomics. Nature Reviews Genetics. 14 Mai 2025