- Accueil >
- Les actualités de l'Institut Curie >
- Comprendre l'architecture et l'organisation des centromères
Les travaux décrits dans cette publication, parue le 22 mars 2022 dans Molecular Cell, fournissent des indications sur l'importance de la topologie de l'ADN pour la régulation et la stabilité des centromères, qui ressemblent à celles d'autres régions répétitives du génome humain, comme les télomères et les transposons.
L'organisation des chromosomes humains est largement étudiée et une attention particulière est accordée au rôle de l'ADN répétitif. De nombreuses recherches se concentrent sur les centromères, les structures chromosomiques bien connues qui guident la ségrégation correcte du génome pendant la mitose et la méïose. Les centromères sont des structures à régulation épigénétique avec des répétitions d'ADN, chez plusieurs espèces. Ces séquences uniques interrogent sur l'architecture et la fonctionnalité des répétitions centromériques.
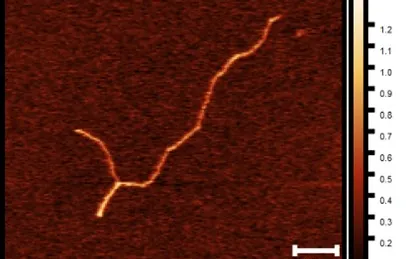
Notre article fournit le premier rapport sur les structures complexes de l'ADN centromérique humain, qui régulent le positionnement et l'intégrité des centromères. Ce travail est un travail d'équipe où nous avons utilisé des approches interdisciplinaires, telles que la biochimie, les techniques de molécules uniques (AFM, nanopore et pinces optiques) et l'imagerie cellulaire, en collaboration avec les leaders mondiaux des méthodes de molécules uniques de l’université d'Amsterdam (l'équipe de G. Wuite) et de l’université de technologie de Delft (l'équipe de C.Dekker) aux Pays-Bas,
explique Daniele Fachinetti, directeur de recherche CNRS et chef de l’équipe Mécanismes moléculaires de la dynamique des chromosomes dans l’unité Biologie cellulaire et cancer (CNRS UMR 144 / Sorbonne Université / PSL) au Centre de recherche de l’Institut Curie.
L’équipe apporte la preuve directe que l'ADN centromérique humain s'auto-organise en structures secondaires d'ADN principalement en épingles à cheveux de taille nanométrique. Ses travaux révèlent que la liaison de la protéine centromérique B (CENP-B) à des séquences uniques, dans les répétitions centromériques humaines, remodèle les centromères. Cette liaison forme des boucles d'ADN de taille submicronique entre les répétitions d'ADN. Ces dernières compactent la chromatine centromérique et favorisent les interactions inter-chromosomes en formant des amas entre chromosomes. C’est une caractéristique importante pour la préservation du positionnement des centromères en interphase et pour le maintien de l'intégrité de l'ADN pendant la traction des microtubules durant la mitose.
Nos résultats ont des implications importantes, non seulement pour notre compréhension de la topologie de l'ADN et de la régulation centromérique, mais aussi pour la stabilité du génome, car les centromères ou les régions qui les entourent sont des points chauds pour les cassures de l'ADN, mais leurs causes sont inconnues,
s’enthousiasme le biologiste moléculaire.
La découverte surprenante de telles structures ouvre la voie à de futurs travaux sur l'intégrité de ces régions, par exemple pendant la réplication. Leur fragilité peut en effet être à la base de réarrangements chromosomiques observés dans plusieurs types de cancer.
La formation de boucles d'ADN révèle également une organisation unique et nouvelle de haut niveau, qui pourrait jouer un rôle dans le maintien de l'identité et de la position des centromères.
Enfin, nous mettons en évidence un nouveau facteur responsable de l'organisation du génome eucaryote : la capacité de CENP-B à rassembler plusieurs centromères. Ceci pourrait avoir une pertinence fonctionnelle pour séparer certains centromères et contrôler le métabolisme de l'ADN,
conclut le chercheur.
► Lire la publication : https://doi.org/10.1016/j.molcel.2022.02.032
CENP-B-mediated DNA loops regulate activity and stability of human
centromeres
Florian Chardon1,#, Aleksandre Japaridze2,#, Hannes Witt3, Leonid Velikovsky1, Camellia
Chakraborty1, Therese Wilhelm1, Marie Dumont1, Wayne Yang2, Carlos Kikuti1, Stephane
Gangnard1, Anne-Sophie Mace1, Gijs Wuite3, Cees Dekker2 and Daniele Fachinetti1,*
1Institut Curie, PSL Research University, CNRS, UMR 144, 26 rue d’Ulm, F-75005, Paris, France
2Department of Bionanoscience, Kavli Institute of Nanoscience Delft, Delft University of
Technology, Van der Maasweg 9, 2629 HZ, Delft, The Netherlands
3Department of Physics and Astronomy, and LaserLaB Amsterdam, Vrije Universiteit
Amsterdam, De Boelelaan 1081, 1081 HV, Amsterdam
#These authors contributed equally to this work
*Corresponding and lead author: daniele.fachinetti@curie.fr