Présentation

Mon laboratoire cherche à comprendre comment l'héritage chromosomique est réalisé avec une telle fidélité dans les cellules de mammifères. Nous étudions les mécanismes génétiques et épigénétiques qui contrôlent la transmission fidèle de notre matériel génétique.
Plus précisément, nous nous intéressons aux centromères, des loci chromosomiques clés nécessaires à la division cellulaire. Nous cherchons à identifier : i) comment les centromères sont établis, ii) les mécanismes qui régissent la fonction des centromères, iii) comment leur intégrité est préservée au cours du cycle cellulaire et iv) le rôle que joue la défaillance des centromères dans l'instabilité du génome.
Nous utilisons des approches de biologie moléculaire et cellulaire combinées à la génétique, la physique et la biochimie.
Twitter: @FachinettiLab
Bluesky: dfachinetti.bsky.social
Le maintien de l'intégrité du génome et d'un caryotype chromosomique correct est crucial pour la santé humaine. En effet, un nombre anormal de chromosomes - une caractéristique appelée aneuploïdie numérique - et des altérations chromosomiques structurelles sont les caractéristiques de nombreux cancers humains.
L'instabilité du génome peut survenir tout au long du cycle cellulaire, à des moments cruciaux comme la réplication du génome ou la division cellulaire. Coordonner une duplication conforme avec la ségrégation des chromosomes est une tâche essentielle pour maintenir un caryotype correct. Le laboratoire Fachinetti s'intéresse à la révélation des processus qui régissent la réplication de l'ADN et la division cellulaire, et à la manière dont ils sont coordonnés. Ceci est primordial pour comprendre comment prévenir l'apparition de l'instabilité du génome et des états pathologiques qui en découlent.
Le stress de réplication est une menace majeure pour l'intégrité du génome et joue un rôle causal important dans le cancer et le vieillissement. On sait que les cassures chromosomiques dépendantes du stress de la réplication ciblent les régions génomiques qui présentent des caractéristiques spécifiques, telles que les régions à réplication tardive, les sites d'initiation réduite de la réplication de l'ADN, les régions qui présentent des niveaux élevés de transcription ou les longues portions d'ADN répétitif, car elles sont plus sujettes à la formation de structures secondaires de l'ADN. Par conséquent, il est essentiel de comprendre la nature des lésions liées à la réplication et la façon dont les cellules réagissent au stress de la réplication pour élucider les événements précoces menant à la tumorigenèse, ainsi que pour développer de nouvelles stratégies de thérapie anticancéreuse.
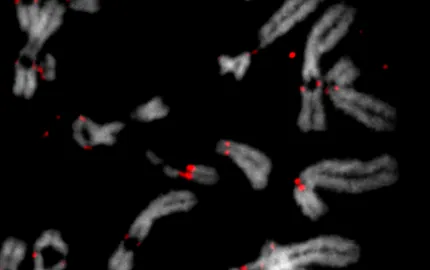
La division cellulaire est une étape essentielle de la vie de toutes les cellules. Lorsqu'une cellule se sépare, elle doit répartir équitablement ses chromosomes, la base moléculaire de l'hérédité génétique, entre ses deux cellules filles (figure and video). Le centromère joue un rôle clé dans ce processus : c'est une petite région sur chaque chromosome qui sert de point d'ancrage pour la fixation de la machinerie cellulaire (le kinétochore) qui délivre une copie de chaque chromosome dupliqué à chaque cellule fille. Une défaillance de ces processus peut entraîner une mauvaise ségrégation des chromosomes (video) et, par conséquent, des altérations numériques et structurelles.
Figure : Les centromères sont nécessaires à la ségrégation des chromosomes au cours de la mitose et de la méiose. L'immunofluorescence sur des cellules humaines montre la localisation de CENP-C et d'alpha-tubuline au cours des différentes étapes de la mitose
Video : Vidéo à intervalles réguliers d'une cellule humaine diploïde, non transformée, de l'épithélium pigmentaire de la rétine (RPE-1) pendant la mitose sans (à gauche) ou avec des événements de mauvaise ségrégation (à droite). L'ADN est en cyan et l'alpha-tubuline en rouge.
Le laboratoire Fachinetti vise à identifier : i) comment les centromères sont constitués, ii) les mécanismes qui contrôlent la fonction des centromères, iii) comment ils sont répliqués et comment leur intégrité est préservée à travers le cycle cellulaire ainsi que iv) le rôle de la défaillance des centromères dans l'instabilité du génome. Nous accordons une attention particulière au rôle de l'ADN centromérique : tous les centromères humains natifs sont constitués de séquences d'ADN répétitives qui s'étendent sur plusieurs mégabases et représentent environ 3 % du génome humain total. Nous supposons que l'ADN centromérique est un élément clé des centromères humains pour maintenir la position et l'assemblage des centromères, qui à leur tour, maintiennent une ségrégation chromosomique fidèle (Dumont, M. & Fachinetti, D. PMSB, 2017 ; Scelfo, A. & Fachinetti, D. Cells, 2019 ; Gamba, R. & Fachinetti, D. ECR, 2020). D'autre part, les répétitions centromériques pourraient agir comme des régions potentiellement fragiles qui, dans certaines conditions, sont plus sujettes à des événements de rupture et de recombinaison (Barra, V. & Fachinetti, D. Nat Com, 2018). Notre programme de recherche est basé sur une approche intégrée qui combine l'utilisation de modèles de culture modifiés (cellules humaines et cellules souches embryonnaires de souris) pour contrôler de manière conditionnelle la stabilité des protéines endogènes via un degron inductible par l'auxine (AID) (Holland, A. J., Fachinetti, D. et al PNAS, 2012 ; Hoffmann, S. & Fachinetti, D. Meth, 2018) avec l'imagerie cellulaire, l'analyse cytogénétique, le séquençage de l'ADN, les approches protéomiques spécifiques à un locus, le criblage à l'échelle du génome et les technologies de molécules uniques sur l'ADN et la chromatine dans les cellules vivantes (Keizer, V. et al, BioRxiv, 2021).
Nous pensons que nos recherches apporteront une vision transformatrice des mécanismes moléculaires à l'origine de la genèse de l'instabilité du génome.
Bibliographie
- Mellone, B* and Fachinetti, D*. (*Co-corresponding authors). Diverse mechanisms of centromere specification. (2021) Curr Biol. Nov 22;31(22):R1491-R1504. https://pubmed.ncbi.nlm.nih.gov/34813757/
- Dumont, M and Fachinetti, D (2017). DNA sequences in centromere formation and function. Book chapter Springer, Centromeres and Kinetochores. Prog Mol Subcell Biol. 56:305-336. https://pubmed.ncbi.nlm.nih.gov/32944612/
- Scelfo, A. and Fachinetti, D. (2019). Keeping the centromere under control: a promising role for DNA methylation. Cells, 16;8(8). https://pubmed.ncbi.nlm.nih.gov/31426433/
- Gamba, R. and Fachinetti, D. From evolution to function: two sides of the same CENP-B coin? (2020) Exp Cell Res. May 15;390(2):111959. https://pubmed.ncbi.nlm.nih.gov/32173469/
- Barra, V. and Fachinetti, D. (2018). The dark side of the centromeres: types, causes and consequences of structural abnormalities implicating centromeric DNA. Nature Communications, 9(1):4340. https://pubmed.ncbi.nlm.nih.gov/30337534/
- Holland, A.J.*, Fachinetti, D.*, Han, J.S., and Cleveland, D.W. (2012). (* Co-first authorship). Inducible, reversible system for the rapid and complete degradation of proteins in mammalian cells. PNAS, 109, E3350-7. https://pubmed.ncbi.nlm.nih.gov/23150568/
- Hoffmann, S and Fachinetti, D (2018). Real-Time De Novo Deposition of Centromeric Histone-Associated Proteins Using the Auxin-Inducible Degradation System. Book chapter, Methods Mol Biol. 2018;1832:223-241. https://pubmed.ncbi.nlm.nih.gov/30073530/
- Gamba, R., Mazzucco, G., Wilhelm, T., Chardon, F., Velikovsky, L., Picotto, J., Doksani, Y. and Fachinetti, D. A method to enrich and purify centromeric DNA from human cellsbioRxiv 2021.09.24.461328; doi: https://doi.org/10.1101/2021.09.24.461328
- Keizer, V., Grosse-Holz, S., Woringer, M., Zambon, L., Aizel, K., Bongaerts, M., Kolar-Znika, L., Scolari, V., Hoffmann, S., Banigan, E., Mirny, L.A., Dahan, M., Fachinetti, D.* and Coulon, A. * (*Co-corresponding authors). Live-cell micromanipulation of a genomic locus reveals interphase chromatin mechanics. bioRxiv 2021.04.20.439763; doi: https://doi.org/10.1101/2021.04.20.439763
























 MEET THE PI
MEET THE PI